
单细胞RNA测序(scRNA-seq)可通过转录谱分析发育、再生、衰老、以及疾病发生过程中的细胞和分子机制。然而,目前大多数的定量方法局限在基因水平,无法区分具有不同结构和功能的转录异构体的差异表达。
最近,我校中山眼科中心刘奕志和胡友金团队开发了一种低成本、高效的新型单细胞测序技术scRCAT-seq,并应用于分析人视网膜发育的细胞图谱,发现视网膜干细胞的分化及细胞命运决定过程中TSS和TES的变化规律。研究成果于2020年10月13日,在Nature Communications在线发表论文“Single-cell RNA cap and tail sequencing (scRCAT-seq) reveals subtype-specific isoforms differing in transcript demarcation”。
结合转录本首尾捕获和机器学习系统分析TSS和TES
在此研究中,scRCAT-seq通过对单细胞全长cDNA的5’及3’进行靶向捕获,基于常规的短读长测序平台进行测序(图1),结合随机森林(random forest, RF)等机器学习算法获得高准确性的TSS和TES(图2)。与已报道的测序方法进行比较,scRCAT-seq可显著提高所鉴定TSSs/TESs的准确性4-5倍。

图1:scRCAT-seq的实验流程示意图


图2:机器学习算法显著提高所鉴定TSSs/TESs的准确性
ScRCAT-seq具有低成本、高效率的特点
与已有的全长转录本测序方法Smart-seq2和ScISOr-seq相比,scRCAT-seq具有成本低,效率高等特点。以相同的成本,scRCAT-seq所能检测的转录本数量是ScISOr-seq的7倍(图3),scRCAT-seq能检测到大量ScISOr-seq所不能检测到的低表达的基因。

图3:scRCAT-seq具有高效率、低成本的特点
scRCAT-seq的可适性强
scRCAT-seq适用于分析不同物种来源、不同细胞类型和数量的样品,进行细胞分群,细胞类型特异的转录本鉴定,稳定性好,并可在不同物种和细胞中标注新的转录本,丰富现有标注RNA转录本的数据库。scRCAT-seq可与单细胞三代测序技术,或者sanger测序技术结合,对全长转录本的结构,特别是内在外显子进行进一步鉴定。
应用于分析人视网膜发育过程中转录组TSS和TES的动态变化
以人胚胎干细胞来源的视网膜类器官为研究对象,通过对10571个单细胞进行测序分析,该研究描绘了视网膜祖细胞(RPC)向光感受器前体细胞(PR precursors),进一步向视锥细胞(cone)分化的转录异构体的表达图谱,发现转录异构体变化最显著的时间点发生在RPC向光感受器前体细胞转变的节点。该研究还发现CCND1异构体可能通过调节细胞周期,介导RPC向光感受器前体细胞的命运转换(图4)。
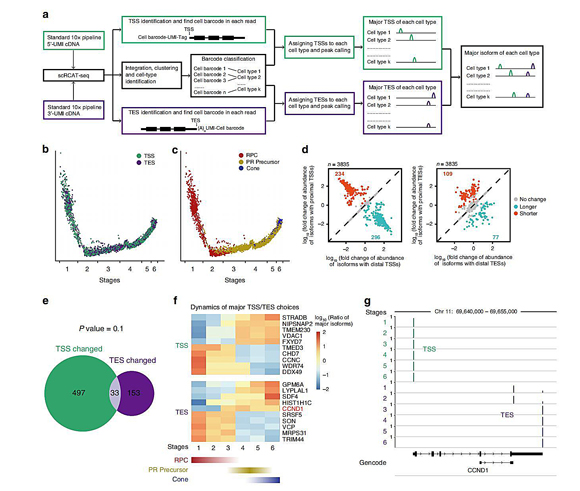
图4:人类视锥细胞发育过程中RNA异构体的动态变化